
IBS 나노의학 연구단, 13종 유전자 가위 교정 효율 비교분석
가장 효율적인 유전자 가위로 최상의 조건에서 질환 치료 기대
유전자를 교정하고자 하는 상황에 따라 가장 효율적인 유전자 가위를 추천해주는 알고리즘이 개발됐다. 기초과학연구원(IBS, 원장 노도영) 나노의학 연구단(단장 천진우) 김형범 연구위원(연세대의대 약리학교실 교수) 연구팀은 13종 유전자 가위 변이체들의 효율을 비교‧분석하고, 표적 염기서열에 따른 최적의 교정 도구를 골라주는 딥러닝 기반 시스템인 ‘DeepSpCas9variants’를 개발했다.
크리스퍼 유전자 가위(CRISPR-Cas9)는 유전자의 특정 지점을 교정하는 기술이다. 유전자 가위는 표적으로 정한 DNA의 특정 염기 서열 정보를 지닌 가이드RNA와 염기서열을 자르는 절단효소로 구성된다. 사람 몸에 자주 침입하는 세균인 화농성연쇄상구균(S.pyogenes)에서 가져온 SpCas9가 절단효소로 가장 널리 쓰인다.
SpCas9는 효율이 높다는 장점이 있지만, 표적(target) 이외의 지점을 잘라내는 표적이탈(off-target)이 빈번히 발생한다는 제한이 있다. 이에 따라 표적이탈 발생 문제를 보완하기 위해 정확성을 높인 변이체(high-fidelity variant) 1)들이 개발됐다. 이와 함께 유전자 가위의 범용성을 높인 PAM 변이체(PAM variant) 2)도 여럿 개발됐다.
PAM 서열 PAM(Protospacer-Adjacent Motif) 3)은 유전자 가위가 절단하려는 표적 DNA를 인식하도록 돕는 일종의 알림판 역할을 한다.
이렇듯 유전자 가위의 약점 보완을 위해 여러 절단효소 변이체가 개발됐지만, 각 성능과 장‧단점을 체계적으로 분석한 연구는 없었다. 이 때문에 어떤 상황에서 어떤 유전자 가위를 사용해야 하는지 선택이 어려웠다.
연구진은 대용량 검증 기술을 이용해 13종 SpCas9 변이체들의 상황에 따른 유전자 교정 효율을 밝혀내 이 문제를 해결했다. 연구진은 동일한 조건에서 인간배아신장세포를 이용해 유전자 교정 실험을 진행하며, 8종의 PAM 변이체의 교정 효율을 분석했다. 그 결과, 4종의 변이체가 인간배아신장세포 교정에 적절한 것으로 나타났다. 그중 범용성이 가장 높은 것으로 평가된 SpCas9-NG 절단효소의 경우 PAM 서열로 쓰일 수 있는 156개의 서열 중 89개의 서열을 인식했다. 다른 변이체에 비해 표적할 수 있는 부위가 더 많다는 의미다.
한편, 6종의 정확성을 높인 변이체 중 표적이탈 발생이 가장 적은 변이체도 찾아냈다. evoCas9가 0.89의 정확성을 보여 가장 높게 평가됐다. 반면, SpCas9의 정확성은 0.35로 나타났다.
이 정보를 토대로 연구진은 최적의 유전자 가위를 추천해주는 딥러닝 기반의 알고리즘인 ‘DeepSpCas9variant’를 개발했다. 이 알고리즘을 이용하면 교정하고자 하는 특정 염기서열을 인식할 수 있는 변이체를 확인할 수 있는 것은 물론 기대되는 교정효율까지 알아낼 수 있다.

김형범 연구위원은 “지금껏 밝혀지지 않았던 여러 유전자 가위 변이체들의 차이를 체계적으로 분석한 연구로, 정확한 유전자 교정 도구를 선택하는 가이드라인을 제시할 수 있다”며 “표적이탈로 인한 돌연변이 등 부작용을 최소화하며 가장 효율적인 도구를 이용해 최상의 조건에서 유전적 질환을 치료할 수 있게 될 것”이라고 말했다.
이번 연구성과는 네이처 바이오테크놀로지(Nature Biotechnology, IF 31.864) 6월 9일자 온라인판에 게재됐다.
1) 정확성을 높인 변이체(High-fidelity variant) : 유전자 가위의 표적이탈 발생 확률을 줄이기 위해 개발된 변이체들로 eSpCas9, SpCas9-HF1, HypaCas9, evoCas9, Sniper-Cas9, xCas9 등이 있다.
2) PAM 변이체(PAM variant) : 유전자 가위가 인식할 수 있는 염기서열을 다양화하기 위해 개발된 변이체들로 VQR, VRER, VRQR, QQR1, xCas9, SpCas9-NG 등이 있다. 한편, VRQR-HF1과 xCas9은 표적이탈 발생 확률과 PAM 범용성을 모두 개선하고자 개발된 절단효소다.
3) PAM(Protospacer-Adjacent Motif) : 유전자 가위가 DNA 절단을 시작하기 위해서는 표적 DNA의 한쪽 말단에 접근해야 하는데, 이때 활용되는 짧은 유전자 염기서열이다. PAM 서열이 없는 곳은 표적으로 인지하지 못하기 때문에 SpCas9의 표적이 될 수 없다.
연구 추가 설명
Prediction of the sequence-specific cleavage activity of Cas9 variants
저자정보
Nahye Kim(제1저자), Hui Kwon Kim(제1저자), Sungtae Lee, Jung Hwa Seo, Jae Woo Choi, Jinman Park, Seonwoo Min, Sungroh Yoon, Sung-Rae Cho and Hyongbum Henry Kim(교신저자)
연구이야기
[연구 배경]
크리스퍼 유전자 가위는 유전자 특정 부위를 절단하여 유전자를 교정하는 인공 제한효소다. 크리스퍼 유전자 가위가 개발된 이후, 이를 이용하여 유전자치료, 질병 모델 구축, 유전자 기능 규명 등 많은 분야에서 폭넓게 쓰이고 있으며, 현재 가장 널리 쓰이고 있는 유전자 가위는 화농성연쇄상구균(Streptococcus pyogenes)에서 유래한 Cas9(SpCas9) 이다.
SpCas9이 표적을 자르기 위해서는 표적에 PAM 서열이 필요하다. PAM 서열이 없을 경우 SpCas9이 인지하지 못하여 표적이 될 수 없기 때문에, SpCas9 사용에 있어 제약으로 여겨진다. 이를 극복하기 위해 SpCas9이 인지하는 PAM 서열을 변경한 여러 PAM 변이체들이 발표되었다. 또한, SpCas9이 효율적으로 표적을 자르는 반면 원치 않는 유전자를 자르는 표적이탈(off-target) 효율이 높아, 정확하게 원하는 유전자만 교정할 수 있도록 SpCas9의 표적이탈을 줄인 여러 high-fidelity variant 들이 발표되었다. 하지만, 지금껏 발표된 여러 가지 variant들 중 어떠한 것을 사용해야하는지에 대해 정확히 비교된 바가 없어 연구자들이 혼선을 빚었다.
[연구 과정]
연구팀은 유전자가위 효율을 대량으로 측정할 수 있는 라이브러리 시스템을 이용하여, 여러 SpCas9 변이체들의 가이드 RNA 표적 염기서열에 따른 교정 효율을 측정하였다. 이를 통해 특정 PAM 서열에서 어떠한 변이체를 사용해야하는지와 교정 정확성 차이를 밝혀내었다. 또한, 해당 데이터를 이용하여 다양한 변이체들에 대한 가이드 RNA의 효율을 예측하는 알고리즘을 개발하여 연구자들이 어떠한 변이체를 사용해야 하는지에 대해 선택할 수 있도록 하였다.
[어려웠던 점]
본 연구는 13가지 종류의 변이체들을 동시 다발적으로 대용량으로 비교하였기 때문에 실험 과정을 모두 동일한 조건으로 유지하는 것이 아주 중요하였다. 서로 다른 종류의 변이체 간 차이가 나지 않도록 동시에 모두 실험하여 실험간 차이를 최소화 하거나, 실험 시간을 동일하게 맞추는 등 여러 가지 실험 조건들을 일정하게 유지하는 것이 쉽지 않았다.
[향후 연구계획]
이번 연구를 통해 구축된 시스템을 기반으로 가장 효율적인 가이드 RNA와 정확하게 원하는 유전자만 교정할 수 있는 변이체를 선정하여 최상의 조건 아래에서 실험할 수 있게 되었다. 선정된 가이드 RNA 및 variant로 다양한 유전적 질환을 교정하는 연구를 진행하여 유전질환 치료에 적용해볼 예정이다.
그림 설명
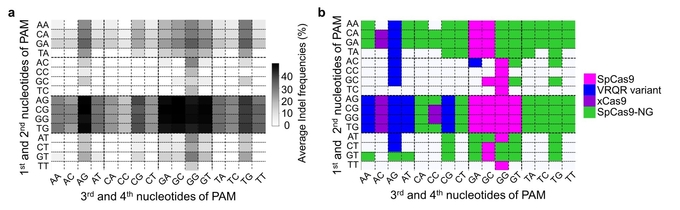
[그림1] PAM 서열에 따른 최적의 유전자 가위
연구진은 PAM 서열에 따라 기대되는 유전자 교정 효율을 분석했다(a). 색이 진할수록 효율이 높다는 의미로 가령, 검은색으로 나타낸 NGAG 서열 등에서 높은 교정 효율을 기대할 수 있다. 또한 특정 PAM에서 가장 높은 유전자 교정 효율을 보이는 PAM 변이체도 찾아냈다(b). 위 그림에서 NGAG 서열의 경우 VRQR 변이체를 사용하는 것이 가장 적합하다.
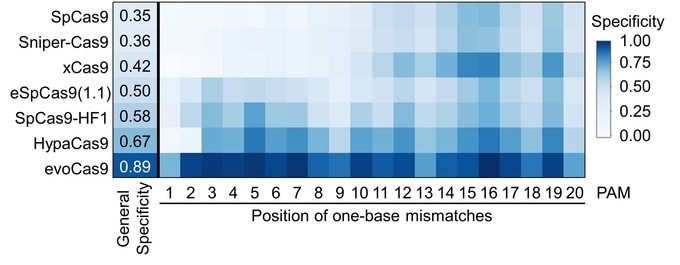
[그림 2] 정확성을 높인 변이체(High-fidelity variant)의 교정 정확도 분석
연구진은 20개의 염기서열로 구성된 가이드RNA에서 1개의 염기서열만 의도적으로 바꾼 뒤(one-base mismatch) 유전자 가위 변이체가 표적을 얼마나 교정하는지를 분석했다. 20개의 염기가 정확하게 일치한 표적을 자르는 경우를 on-target, 100% 일치하지 않는 표적을 자르는 경우를 off-target이라고 한다. 그 결과, evoCas9이 0.89로 가장 높은 정확성을 나타냈다. 표적이탈 발생이 가장 적다는 의미다.
연구진 이력사항

[김형범 IBS 나노의학 연구단 연구위원, 교신저자>]
1. 인적사항
· 소속 : IBS 나노의학 연구단,
연세대학교 의과대학 약리학교실
· 전화 : 02-2228-0879
· 이메일 : hkim1@yuhs.ac
· 1995 – 2001 연세대학교 의과대학 학사 졸업
· 2001 – 2003 연세대학교 의과대학 석사 졸업
· 2003 – 2006 연세대학교 나노과학기술협동과정 박사 졸업
3. 경력사항
· 2007 – 2010 Postdoctoral Fellow; Emory/Tufts University
· 2010 – 2011 차의과대학교 조교수
· 2011 – 2015 한양대학교 조교수/부교수
· 2015 – 현재 연세대학교 의과대학 약리학교실 조교수/부교수/교수
· 2016 – 현재 IBS 나노의학 연구단 연구위원

[김나혜 연세대 의과대학 학생연구원, 제1저자]
1. 인적사항
· 소속 : 연세대학교 의과대학 약리학교실
· 이메일 : wnstn_1259@hanmail.net
· 2013.03 – 2017.02 성균관대학교 생명과학과 학사 졸업
· 2017.03 - 현재 연세대학교 의과대학 의과학과 통합과정

[김희권 IBS 나노의학 연구단 박사후연구원, 제1저자]
1. 인적사항
· 소속 : IBS 나노의학 연구단,
연세대학교 의과대학 의과학과/약리학교실
· 이메일 : huikwonkim@yuhs.ac
· 2009 – 2013 한동대학교 생명과학부 학사 졸업
· 2013 – 2015 한동대학교 생명과학부 석사 졸업
· 2015 – 2020 연세대학교 의과대학 의과학과 박사 졸업
3. 경력사항
· 2020.03–현재 박사후연구원; IBS 나노의학 연구단, 연세대학교 의과대학 의과학과/약리학교실
June 27, 2020 at 07:09PM
https://ift.tt/3dyDnMU
최적 효율의 유전자 가위 추천 알고리즘 개발 - 이엠디
https://ift.tt/37lItuB
0 Comments:
Post a Comment